基因衝突塑造生殖與染色體的快速演化
我們的研究關注於內部基因衝突(Intrinsic genetic conflicts)如何驅動必需基因與基因體結構的演化。一般而言,生存與生殖必需的基因應該高度保守;然而,部分基因卻出現快速演化,甚至在特定譜系中退化。同樣地,染色體的融合與易位通常為有害的突變,但染色體結構在不同物種間變化極大,並常在物種形成與複雜性狀的演化中扮演關鍵角色。
我們的研究建立在一個假設:這些出人意料的演化模式是由內部基因衝突所驅動,即自私遺傳因子(如減數驅動子與轉座子)與宿主基因體之間的演化軍備競賽。這些衝突會在基因體中不僅在基因上留下分子痕跡,同時也能刺激生物演出不同的功能,進而重塑染色質、調控網絡與染色體結構。
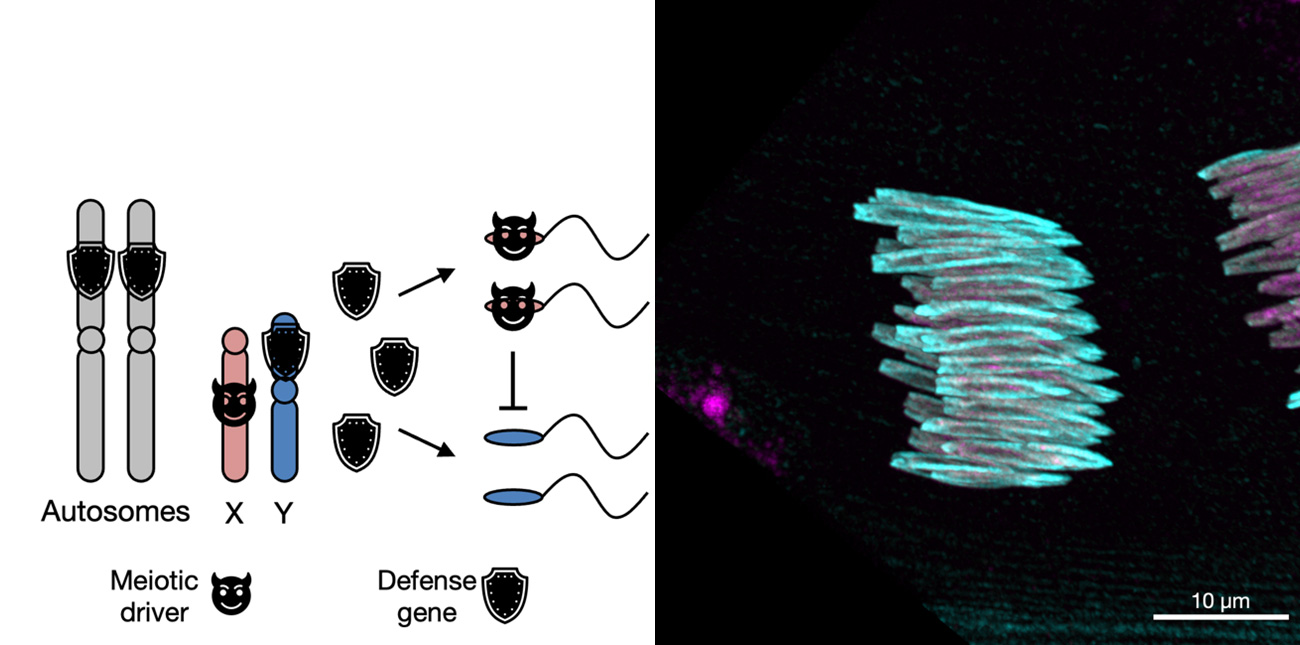
我們的實驗室致力於研究減數驅動子(meiotic drivers):一類會透過淘汰未攜帶它們的配子來偏向自身傳遞至下一代的自私遺傳因子。儘管這些元素會降低生育力,它們仍能迅速在族群中擴散,迫使宿主基因體演化出對抗機制。我們的研究已鑑定並分析了一群可能在抑制跨物種的減數驅動現象中扮演關鍵角色的快速演化必需基因。在這些基因中,我們特別聚焦於精子核蛋白(protamines),不同於其他細胞,精子此用精子核蛋白取代組蛋白(Histones)來纏繞DNA並對雄性生殖功能至關重要。透過解析減數驅動的分子機制,我們希望了解為何以及如何雄性生殖系統會在各物種中快速的演化。
我們也正在研究一類非典型染色體,包括性染色體、只存在於生殖細胞的染色體(germline-restricted chromosomes)與 B 染色體。這些的染色體違反孟德爾遺傳規律,常演化出透過調控染色體分離或細胞分裂的機制,來自私地增加自身傳遞到下一代的機率。藉由探究它們的特殊行為,我們期望理解染色體在非孟德爾遺傳情境下如何演化,以及生物體如何容忍或對抗這些非典型染色體並維持基因體完整性。
我們的研究目標是揭示生殖與染色體生物學的基本原則,並提供對染色體不穩定與重組機制的深入理解,特別是那些與疾病與癌症相關的現象。
- PDF, 2020-2025, Division of Basic Sciences, Fred Hutchinson Cancer Center
- Ph.D., 2014-2020, Department of Biology, University of Rochester
- M.S., 2009–2011, Institute of Ecology & Evolutionary Biology, National Taiwan University
- BS, 2005-2009, Department of Life Science, National Taiwan University
- Zakerzade R#, Chang C-H#, Chatla K, Krishnapura A, Appiah SP, Zhang J, Unckless RL, Blumenstiel JP, Bachtrog D, Wei KH* (2024) Diversification and recurrent adaptation of the synaptonemal complex in Drosophila. PLoS Genet 21(1): e101154
- Chang C-H*, Mejia Natividad I% and Malik HS (2023) Genetic conflicts between sex chromosomes drive expansion and loss of sperm nuclear basic protein genes in Drosophila. eLife 12:e85249.
- Chang C-H*, Gregory EL%, Gordon KE, Meiklejohn CD and Larracuente AM* (2022) Unique structure and positive selection promote the rapid divergence of Drosophila Y chromosomes. eLife 11:e75795
- Chakraborty M#, Chang C-H#, Khost D, Vedanayagam J, Adrion JR, Liao Y, Montooth KL, Meiklejohn CD, Larracuente AM*, and Emerson JJ* (2021). Evolution of genome structure in the Drosophila simulans species complex. Genome Research, 31, 380-396.
- Chang C-H#, Chavan A #, Palladino J#, Wei X, Martins NMC., Santinello B, Chen CC, Erceg J, Beliveau BJ, Wu CT, Larracuente AM*, and Mellone BG* (2019). Islands of retroelements are major components of Drosophila centromeres. PLoS Biology, 17(5), e3000241. (F1000 recommended)
- Chang C-H*, and Larracuente AM* (2019). Heterochromatin-Enriched Assemblies Reveal the Sequence and Organization of the Drosophila melanogaster Y Chromosome. Genetics, 211(1), 333-348. (Highlight article, F1000 recommended)
- Chang C-H*, and Larracuente AM (2017). Genomic changes following the reversal of a Y chromosome to an autosome in Drosophila pseudoobscura. Evolution, 71(5), 1285-1296.