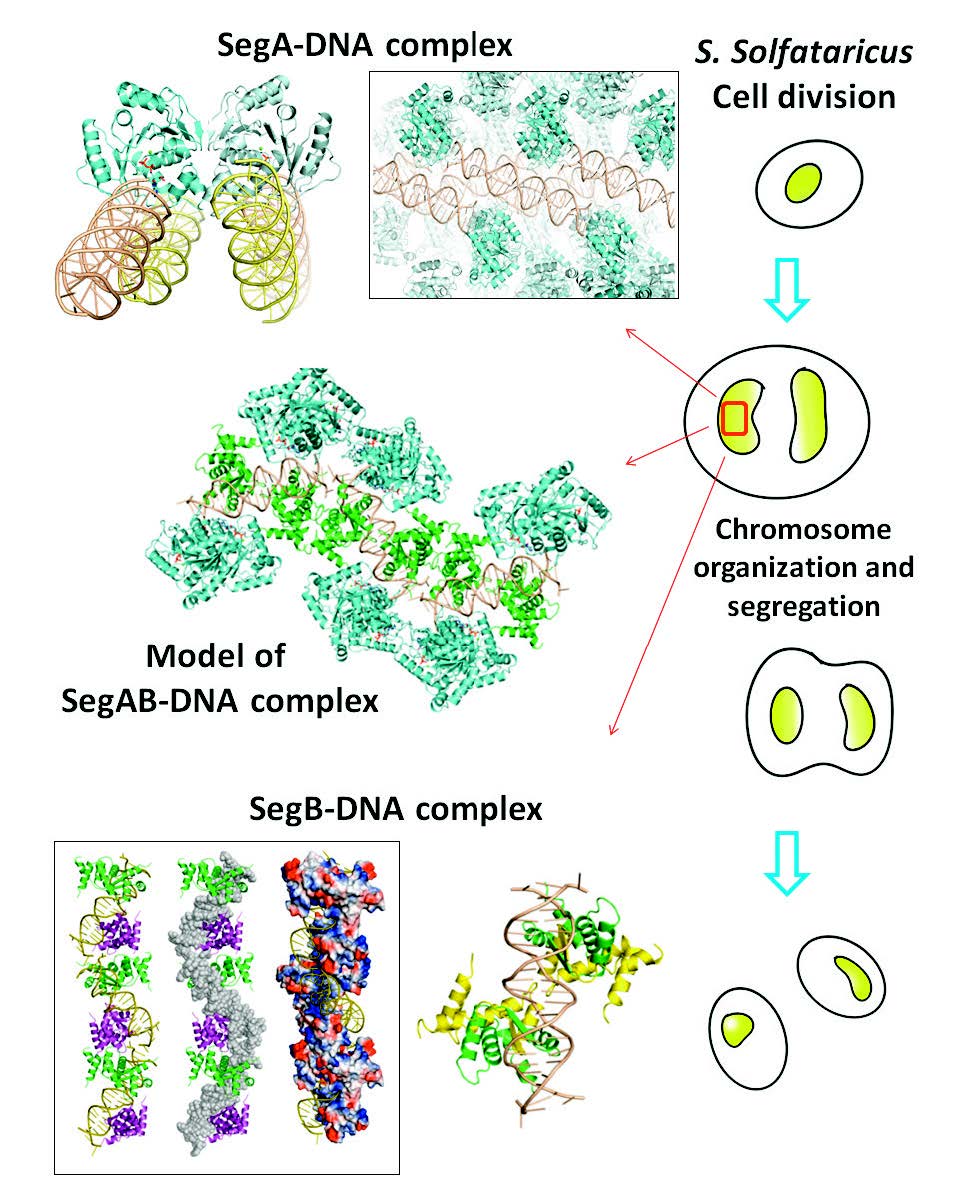
染色體分離的結構與機制研究
生物體的繁殖必需經過遺傳物質之複製且複製後之染色體DNA 須進一步的分配或分離,藉以確保遺傳物質被正確地保留。 在真核生物中,複製後的染色體係通過有絲分裂之紡錘體進行分離,不同於真核生物,細菌染色體分離主要是受 ParABS 系統所調控。不論是真核亦或是原核生物,其染色體分離的過程皆有著廣泛且詳細的了解。 然而,獨立於真核和原核生物外第三種生命型態「古細菌(archaea)」,其染色體分離之詳細機轉仍尚未釐清且缺乏相關資訊。近期的研究成功定義出兩個參與於古細菌染色體分離之相關蛋白,SegA 與 SegB。然而,目前的研究對 SegA 和 SegB 仍然缺乏蛋白質結構以提供更深入的資訊去釐清詳細的古細菌染色體分離機轉。 在此計畫中我們將結合各項生物功能與生物物理之技術以探討 SegA 與 SegB 之間的相互關係。希望藉由不同的成員的組合提供更多的細節,以期能夠更深入的了解古細菌染色體分離的過程與詳細的機轉。此計畫的實驗結果將提供染色體分離中缺失的板塊,並闡述古細菌染色體分離確切之機轉。
參與DNA複製重新起始受損複製叉的蛋白質結構與功能研究
在所有生物體中,遺傳信息的精確和可靠的複製是維繫生存所必要的。然而,當細胞遭受UV、環境中不同因子的壓力亦或是其他傷害時,常會造成DNA 模板損傷而使得DNA 複製過程中斷,細胞為了存活必須有重新活化複製過程的機制。 在細菌中,這個機制被稱為「DNA 複製重啟」,這整個過程是被一群統稱為引物合成體(primosome)的蛋白質所調控。因此對於細菌而言,從新起始修補後的複製叉成為主要的維持生命的後勤程序。 將基因合成所需的整個蛋白組重新集合於受損的基因上需要結合參予在基因複製、重組、修補三方面的各種蛋白,因此提供一個欲維持基因體正常功能所需的各種方面的連結。 在革蘭氏陽性細菌中,依賴於 PriA 的途徑是主要的複制重啟機制,並且需要引子合成體的成員 PriA,DnaD,DnaB,DnaC/I 複合體。在這個題目中,我們將研究這些蛋白質的結構和功能以及他們之間相互作用的機制。
- PDF, 1993-1995, IMB, Academia Sinica
- Ph.D., 1993, Dept.Crystallography, Univ. of Pittsburgh, USA
- MS, 1984, Dept.Chemistry, Natl. Taiwan Univ.
- BS, 1982, Dept. Chemistry, Chung-Yuan Christian Univ.
- 2000, 中研院年輕學者研究成果獎
- 2002, 科技部傑出獎
- 2007-2011, 中研院深耕計畫
- 2021, 中研院特優研究員獎
- Sun, Y.-J., Forouhar, F., Li, H.-M., Tu, S.-L., Kao, S., Shr, H.-L., Chou, C.-C., Hsiao, C.-D. (2002) Crystal structure of pea Toc34 - a novel GTPase of the chloroplast protein translocon. Nature Structure Biology 9: 95-100.
- Yeh, Y.H., Lin, T., Lin, C.Y. Hsiao, C.D. (2014) Crystal structure and functional characterizations of Ybr137w from Saccharomyces cerevisiae. Mol. Cell Biol. 34: 4500-4512.
- Lou, Y.C., Weng, T.H., Li, Y.C., Kao, Y.F., Chou, S.H., *Hsiao, C.D., *Chen, C. (2015) Structure and dynamics of the polymyxin-resistance-associated response regulator PmrA in complex with the promoter DNA. Nature Communications 6: 8838 doi:10.1038/ ncomms9838.
- Li, Y.C., Naveen, V., Lin, M.G.,Hsiao, C.D. (2017) Structural analyses of the bacterial primosomal protein DnaB reveal that it is a tetramer and forms a complex with a primosomal re-initiation protein. J. Biol. Chem. 292(38): 15744-15757.
- Chu, C.H., Yen, C.Y., Chen, B.W., Lin, M.G., Wang, L.H., Tang, K.Z., *Hsiao, C.D., *Sun, Y.J. (2019) Crystal structures of HpSoj–DNA complexes and the nucleoidadaptor complex formation in chromosome segregation. Nucleic Acids Res. 47(4): 2113-2129.
- Tsai, J.Y., Chu, C.H., Lin, M.G., Chou, Y.H., Hong, R.Y., Yen, C.Y., *Hsiao, C.D., *Sun, Y.J. (2020) Structure of the sodium-dependent phosphate transporter reveals insights into human solute carrier SLC20. Science Advances 7: 6(32): eabb4024.